Sobre a variabilidade do DNA satélite pBuM189 entre populações sul-americanas de Drosophila buzzatii
Tradução automática
O artigo original está escrito em EN language (link para lê-lo) .
O DNA satélite pBuM189 foi analisado em populações de Drosophila buzzatii que cobrem a maior parte da distribuição da espécie na América do Sul. Este satDNA consiste em monômeros ricos em A+T de 189 pb e estudos anteriores mostraram uma rápida taxa de mudança evolutiva deste componente do genoma de D. buzzatii. Um total de 63 unidades de repetição pBuM189 de 14 populações de D. buzzatii (9 do Brasil e 5 da Argentina) foram estudadas. A variabilidade nucleotídica média entre as 63 repetições é de 4,2%. Pelo menos uma repetição (Juan/4) parece ser parte de outra subfamília de satDNA pBuM189. O alinhamento nucleotídico de todas as 63 repetições não revelou substituições nucleotídicas específicas, ou indels, que pudessem discriminar cada população ou grupos de populações geograficamente próximas. Essa falta de diferenciação interpopulacional de satDNA é congruente com dados anteriores de mtDNA que indicam um alto fluxo gênico e muito pouca diferenciação populacional ao longo da maior parte da distribuição de D. buzzatii na América do Sul. O fluxo gênico pode ter sido possível durante eventos de glaciação no Pleistoceno, como o que ocorreu entre 13.000 e 18.000 anos atrás, quando D. buzzatii provavelmente tinha uma distribuição mais contínua do que a observada hoje.
A mosca cactofílica Drosophila buzzatii (buzzatii cluster, repleta group) tem uma ampla distribuição na América do Sul, sendo encontrada na maior parte dos territórios da Argentina, Bolívia, Paraguai e Brasil. A espécie utiliza tecidos em decomposição de várias cactáceas do gênero Opuntia spp. e cactos colunares para se alimentar e reproduzir (PEREIRA et al. 1983). Durante os últimos duzentos anos, os cactos Opuntia foram transportados pelo homem para várias regiões do mundo, incluindo países da bacia do Mediterrâneo, África e Austrália. Consequentemente, D. buzzatii migrou com seus hospedeiros. Essa situação fez de D. buzzatii um organismo apropriado para abordar questões relacionadas a mudanças genéticas durante o processo de colonização (FONTDEVILA et al. 1982; HALLIBURTON e BARKER 1993; ROSSI et al. 1996; FRYDENBERG et al. 2002).
De acordo com dados de inversão cromossômica, o Chaco argentino é o centro mais provável de sua radiação (WASSERMAN 1962; CARSON e WASSERMAN 1965). D. buzzatii também é muito abundante no Chaco, associado a várias espécies diferentes de cactos (Vilela et al. 1980). Em geral, as populações brasileiras estão distribuídas de forma fragmentada em pequenas áreas de vegetação xerofítica, onde foram encontradas em frequências muito baixas (VILELA et al. 1983; TIDON-SKLORZ et al. 1994; TIDON-SKLORZ e SENE 1995). A única exceção é o sul do Brasil, onde populações relativamente grandes foram encontradas (VILELA et al. 1983; RUIZ et al. 2000).
Foi postulado que D. buzzatii colonizou o Brasil através de mudanças na distribuição da vegetação árida durante eventos de glaciação, como os que ocorreram no final do Pleistoceno (BARKER et al. 1985; FIGUEIREDO e SENE 1992).
BAIMAI et al. (1983) analisaram os cromossomos em metáfase de várias populações de D. buzzatii da Argentina e do Brasil. Todas as populações amostradas exibem a mesma placa de metáfase. BARKER et al. (1985) não encontraram diferenças alozimáticas significativas entre as populações sul-americanas. Apenas uma pequena diferenciação local parece ter ocorrido nas populações brasileiras da Bahia (nordeste) e Tramanda´1 (sul). Em outro estudo, FIGUEIREDO e SENE (1992) relataram que das 16 inversões cromossômicas detectadas na Argentina, apenas duas estavam presentes nas populações brasileiras e nenhuma foi encontrada no nordeste do Brasil. ROSSI et al. (1996) e DE BRITO et al. (2002) encontraram altos níveis de uniformidade de mtDNA entre as populações sul-americanas de D. buzzatii e vários testes indicaram alto fluxo gênico ao longo da maior parte da distribuição geográfica de D. buzzatii . DE BRITO et al. (2002) também calcularam que D. buzzatii está presente no Brasil há pelo menos 100 mil anos, sugerindo uma expansão pré-holocênica das populações argentinas de D. buzzatii em direção ao Brasil.
Para obter mais informações sobre a variabilidade genética de D. buzzatii na América do Sul, procuramos um DNA nuclear. O DNA satélite (satDNA) é uma classe de DNA não codificante altamente repetitivo que está organizado em tandem em grandes arranjos homogêneos no genoma de quase todos os organismos eucarióticos. A taxa geralmente rápida de mudança evolutiva das sequências de satDNA é apoiada por várias famílias de satDNA que se mostraram específicas para espécies (BACH-MANN et al. 1989; KING e CUMMINGS 1997).
Estudos anteriores mostraram que as sequências de satDNA podem ser úteis na discriminação de populações conspecíficas (ELDER e TURNER 1994; WU et al. 1999).
KUHN et al. (1999) descreveram o DNA satélite pBuM189 de D. buzzatii. Consiste em unidades de repetição ligeiramente ricas em AT e com 189 pb de comprimento. As cinco sequências pBuM189 descritas foram obtidas do DNA genômico de apenas uma população de D. buzzatii. Experimentos de hibridização e PCR não conseguiram detectar sequências pBuM189 no genoma de espécies intimamente relacionadas, como D. serido, D. borborema ou D. koepferae. Portanto, os dados disponíveis apontam para uma taxa rápida de mudança evolutiva deste componente do DNA nuclear de D. buzzatii.
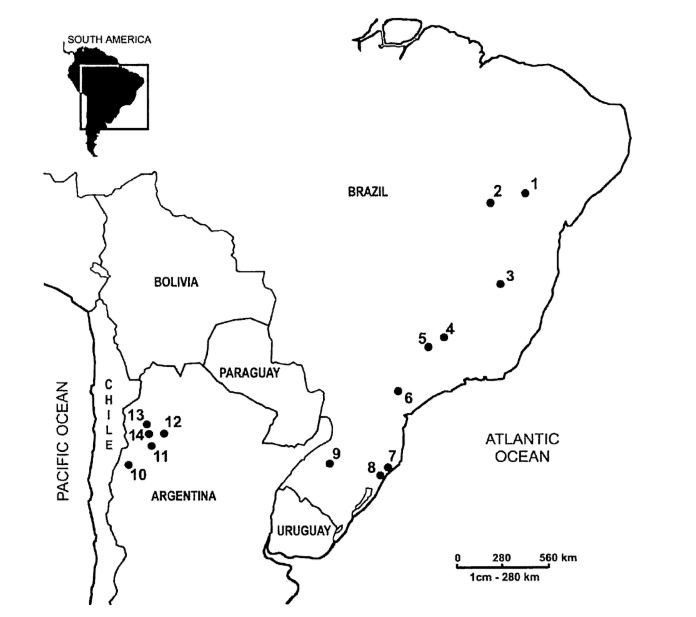
No presente estudo, analisamos 63 sequências de pBuM189 de cepas representando 14 D. buzzatii populações (9 brasileiras e 5 argentinas). As populações cobrem a maior parte da distribuição geográfica desta espécie na América do Sul.
Material e métodos
Amostras
A localização geográfica de todas as populações de D. buzzatii amostradas é mostrada na Fig. 1. As cepas utilizadas com suas respectivas localidades são: D69R5 (1); J79H41 (2); D54F5 (3); H86G8 (4); D42F2 (5); H99X6 (6); H42F1 (7); J25A20 (8); J28E15 (9); ArgE3 (10); Cat (11), Hondo (12); Salta (13) e Ticucho (14).
Técnicas moleculares
As sequências pBuM189 foram originalmente isoladas do genoma de D. buzzatii após a digestão do DNA com a enzima MspI (KUHN et al. 1999). No presente trabalho, a enzima SspI (que também está presente apenas uma vez por unidade de repetição pBuM189) foi utilizada para facilitar o procedimento de clonagem. Fragmentos de satDNA pBuM189 foram identificados pela sua aparência como bandas proeminentes de aproximadamente 190 bp no borrão de fundo do DNA genômico. O DNA da banda de 190 bp foi eluído do gel por incubação noturna em 500mM NaAc; 1mM EDTA. Os fragmentos recuperados foram ligados a um vetor plasmidial pUC18 e transformados em células competentes de Escherichia coli DH5-a. Clones recombinantes foram selecionados como colônias brancas em placas de ampicilina contendo X-gal (5-bromo-4-cloro-3-indolil-β-D-galactosídeo) e IPTG (isopropil-β-D-tiolagalactopiranosídeo). A reação de DNA modelo para sequenciamento foi preparada de acordo com o manual do BigDye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). O sequenciamento automático de DNA foi realizado em um sequenciador ABI Prism™ 377 (Perkin-Elmer).
O programa MEGA 2.1 (KUMAR et al. 2001) foi utilizado para a estimativa da variabilidade nucleotídica, distâncias genéticas e construção de um dendrograma NJ.
Resultados e discussão
Como já era esperado a partir dos dados da sequência pBuM189 anteriores, a digestão do DNA genômico de 13 D. buzzatii cepas com SspI produziu uma banda de DNA fortemente corada de aproximadamente 190 pb, correspondente aos monômeros de pBuM189, seguida por bandas de intensidade decrescente de 380 pb (dímeros), 570 pb (triméros) e assim por diante. A banda de DNA de 190 pb de cada digestão foi eluída independentemente do gel e clonada. Um total de 58 clones foram sequenciados, 39 de 8 populações brasileiras (localidades 1 – 4, 6 – 9) e 19 de 5 populações argentinas (localidades 10 – 14). O alinhamento nucleotídico desses 58 clones com os 5 repetições de pBuM189 (sert/1 – 5 no presente trabalho) da localidade (5) descrita anteriormente por KUHN et al. (1999) provou que todos representam monômeros de satDNA pBuM189 (Fig. 2).

Os 63 repetições pBuM189 são ligeiramente ricas em A+T (71 %) e a variabilidade média da sequência é de 4,2 %. A principal fonte de variação entre as 63 repetições pBuM189 são as substituições de nucleotídeos únicos, sendo 69 % específicas da repetição e o restante comum a duas ou mais repetições. Apenas 11 indels foram encontrados.
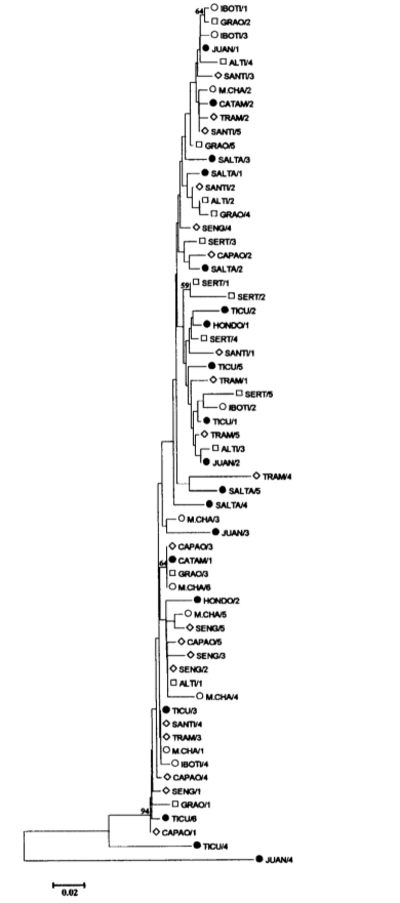
Uma repetição pBuM189 parece ser excepcional. O clone Juan/4 da localidade (10) apresentou níveis inesperadamente altos de divergência de sequência em todas as comparações par a par (22,1 % em média e sempre excedendo 19 %). Além disso, enquanto as sequências típicas de pBuM189 apresentam em média 1,7 % de substituições de nucleotídeos específicas da repetição, 30 foram encontradas na repetição Juan/4 (por exemplo, T nas posições 11 e 42). Tal alto grau de divergência de nucleotídeos sugere que a repetição Juan/4 pode pertencer a outra subfamília de pBuM189. Do ponto de vista teórico, a origem das subfamílias de satDNA depende de variáveis como número de cópias, distribuição genômica da família de satDNA e as taxas relativas de trocas não recíprocas dentro e entre cromossomos não homólogos (DOVER 1986).
Se o repetido Juan/4 for excluído da amostra, a variabilidade nucleotídica média das 62 sequências restantes de pBuM189 diminui para 3,7 %. Este valor é quase idêntico ao 3,8 % calculado por KUHN et al. (1999) com base em cinco sequências de pBuM189 isoladas de apenas uma população de D. buzzatii (localidade 5).
Entre os outros repetições de pBuM189, Ticu/4 da localidade (14) é a mais semelhante a Juan/4 por apresentar dois nucleotídeos específicos (G na posição 70 e A na posição 88) e um indel (posição 49) que são exclusivos do repetido Juan/4 e várias outras substituições em comum com Juan/4 e outros repetições (por exemplo, T na posição 36 ou A na posição 133). O repetido Ticu/4 em si é o repetido pBuM189 mais divergente (10,2 % em média) após Juan/4, apresentando 6 substituições nucleotídicas específicas de repetição (posições 1, 69, 79, 97, 98, 167 e 188). Portanto, o Ticu/4 deve ser considerado como um passo evolutivo intermediário entre os repetições de pBuM189 "típicos" e o altamente divergente repetido Juan/4. Curiosamente, tanto os raros repetições Juan/4 quanto Ticu/4 são da Argentina, onde a espécie provavelmente se originou (WASSERMAN 1962; CARSON e WASSERMAN 1965).
De acordo com dados teóricos e empíricos, a diferenciação das sequências de satDNA deve ocorrer de acordo com o conceito de evolução concertada (DOVER 1986; ELDER e TURNER 1994). Em outras palavras, os arranjos de satDNA de diferentes populações ou espécies podem ser homogeneizados por diferentes mutações devido ao efeito de mecanismos moleculares, como crossing-over desigual e conversão gênica, acoplados ao isolamento genético e à deriva genética.
O alinhamento de nucleotídeos dos 63 repetições de pBuM189 de 14 populações de D. buzzatii (4,5% de repetições em média por população) não revelou nenhuma substituição de nucleotídeo específica, ou indel, que pudesse discriminar uma população específica ou grupos de populações geograficamente próximas (Fig. 2). Em alguns casos, mais de dois clones compartilharam sequência de nucleotídeos idêntica, como no caso dos clones Capao/3, Grao/3, M.Cha/6 e Catam/1 ou M.Cha/1 e Santi/4, mas eles são derivados de localidades geográficas bastante distantes. Por outro lado, nenhuma repetição idêntica foi encontrada em uma única população.
As distâncias genéticas entre as 63 sequências de nucleotídeos pBuM189 foram calculadas de acordo com o método de dois parâmetros de Kimura (KIMURA 1980) e um dendrograma de "vizinho-união" (SAITOU e NEI 1987) foi construído (Fig. 3). A sequência Juan/4 foi localizada em um ramo claramente separado dos outros repetições pBuM189, apoiando a suposição de que pertence a outra subfamília de satDNA pBuM189. A repetição Ticu/4 foi localizada em um ramo entre a Juan/4 e as 61 repetições pBuM189 restantes. Dentro das 61 repetições pBuM189 restantes, nenhum agrupamento contendo sequências pBuM189 de uma população ou grupos de populações geograficamente próximas pôde ser observado.
No total, os dados indicam altos níveis de similaridade da sequência pBuM189 entre populações de D. buzzatii que estão localizadas, em alguns casos, a mais de 3000 km de distância. Essa falta de diferenciação interpopulacional de satDNA é congruente com os dados de mtDNA que indicam um alto fluxo gênico e muito pouca subestrutura populacional ao longo da maior parte da distribuição de D. buzzatii na América do Sul (ROSSI et al. 1996; DE BRITO et al. 2002). Assim, deve haver uma explicação adaptativa para a perda de polimorfismo de inversão cromossômica nas populações brasileiras (comparadas às populações argentinas), conforme relatado por FIGUEIREDO e SENE (1992).
Atualmente, o Chaco (norte da Argentina e leste da Bolívia) e a Caatinga (nordeste do Brasil) são as únicas duas áreas extensas de vegetação árida na América do Sul, apresentando alta densidade e diversidade de espécies de cactos. Em outras áreas, as cactáceas são menos frequentes e distribuídas de forma esparsa. O mesmo é verdadeiro para as populações de D. buzzatii, que dependem de cactos para alimentação e reprodução. É amplamente aceito que o clima global passou por ciclos recorrentes de clima frio-seco e quente-muito durante o Pleistoceno, quando ocorreram pelo menos quatro grandes eventos glaciares e muitos menores. Por exemplo, no último evento de glaciação (de 18.000 a 13.000 anos atrás), o clima na América do Sul era seco e o Chaco estava conectado às Caatingas através de formações xerofíticas no centro do Brasil e na costa atlântica (AB'SABER 1977). Consequentemente, D. buzzatii provavelmente tinha uma distribuição muito mais contínua do que a observada hoje. Essa situação pode ter permitido um grande grau de fluxo gênico entre as populações de D. buzzatii na América do Sul.
Até o momento, a maioria dos DNAs satélites descritos na literatura foi isolada de indivíduos de uma única cepa ou localidade. Os dados presentes são um dos poucos exemplos que fornecem informações sobre a variabilidade da sequência de satDNA entre populações naturais de uma espécie amplamente distribuída.
Autores: Gustavo C. S. Kuhn, Fernando F. Franco, Wilson A. Silva Jr, Nilce M. Martinez-Rossi e Fabio M. Sene
Referências:
- Ab'Saber, N. A. 1977. Espaços ocupados pela expansão dos climas secos da América do Sul, por ocasião dos períodos glaciais quaternários. – Paleoclimas 3: 1 – 19.
- Bachmann, L., Raab, M. e Sperlich, D. 1989. DNA satélite e especiação: um DNA satélite específico de espécie de Drosophila guanche. – Z. Zool. Syst. Evol. Forsch. 27: 84 – 93.
- Baimai, V., Sene, F. M. e Pereira, M. A. Q. R. 1983. Heterocromatina e diferenciação cariotípica de algumas espécies de cactos neotropicais do grupo Drosophila repleta. – Genetica 67: 81 – 92.
- Barker, J. S. F., Sene, F. M., East, P. P. et al. 1985. Polimorfismo alozímico e cromossômico de Drosophila buzzatii no Brasil e na Argentina. – Genetica 67: 161 – 170.
- Carson, H. L. e Wasserman, M. 1965. Um polimorfismo cromossômico amplamente distribuído em uma espécie amplamente distribuída, Drosophila buzzatii. – Am. Nat. 99: 111 – 115.
- De Brito, R. A., Manfrin, M. H. e Sene, F. M. 2002. Filogeografia do DNA mitocondrial de populações brasileiras de Drosophila buzzatii. – Genet. Mol. Biol. 2: 161 – 171.
- Dover, G. A. 1986. Impulso molecular em famílias multigênicas: como novidades biológicas surgem, se espalham e são assimiladas. – Trends Genet. 2: 159 – 165.
- Elder, F. J. e Turner, B. J. 1994. Evolução concertada a nível populacional: sequências de DNA satélite HindIII de peixes pupfish. – Proc. Natl Acad. Sci. 91: 994 – 998.
- Figueiredo, V. L. C. e Sene, F. M. 1992. Variabilidade cromossômica em populações brasileiras de Drosophila buzzatii (Diptera, Drosophilidae). – Revta. Bras. Biol. 52: 555 – 561.
- Fontdevila, A., Ruiz, A., Ocana, J. et al. 1982. A história evolutiva de Drosophila buzzatii. II. Quanto o polimorfismo cromossômico mudou na colonização? – Evolution 36: 843 – 851.
- Frydenberg, J., Pertoldi, C., Dahlgaard, J. et al. 2002. Variação genética em populações originais e colonizadoras de Drosophila buzzatii analisadas por locos de microssatélites isolados com um novo método de triagem PCR. – Mol. Ecol. 11: 181 – 190.
- Halliburton, R. e Barker, J. S. F. 1993. Falta de variação do DNA mitocondrial em Drosophila buzzatii australiano. – Mol. Biol. Evol. 10: 484 – 487.
- Kimura, M. 1980. Um único método para estimar a taxa evolutiva de substituições de bases através de estudos comparativos de sequências nucleotídicas. – J. Mol. Evol. 16: 111 – 120.
- King, L. M. e Cummings, M. P. 1997. A variação da sequência de repetição do DNA satélite é baixa em três espécies de besouros enterradores do gênero Nicrophorus (Coleoptera: Silphidae). – Mol. Biol. Evol. 14: 1088 – 1095.
- Kuhn, G. C. S., Bollgönn, S., Sperlich, D. et al. 1999. Caracterização de um DNA satélite específico de espécie de Drosophila buzzatii. – J. Zool. Syst. Evol. Res. 37: 109 – 112.
- Kumar, S., Tamura, K., Jakobsen, I. B. et al. 2001. MEGA2: software de análise genética evolutiva molecular. – Arizona State Univ.
- Pereira, M. A. Q. R., Vilela, C. R. e Sene, F. M. 1983. Notas sobre locais de reprodução e alimentação de algumas espécies do grupo repleta do gênero Drosophila (Diptera, Drosophilidae). – Ciência e Cultura 35: 1313 – 1319.
- Rossi, M.S., Barrio, E., Latorre, A. et al. 1996. A história evolutiva de Drosophila buzzatii. XXX. Polimorfismo do DNA mitocondrial em populações originais e colonizadoras. – Mol. Biol. Evol. 13: 314 – 323.
- Ruiz, A., Cansian, A. M., Kuhn, G. C. S. et al. 2000. O quebra-cabeça da especiação de Drosophila serido: juntando novas peças. – Genetica 108: 217 – 227.
- Saitou, N. e Nei, M. 1987. O método de vizinho-união: um novo método para reconstruir árvores filogenéticas. – Mol. Biol. Evol. 4: 406 – 425.
- Tidon-Sklorz, R. e Sene, F. M. 1995. Fauna de Drosophila (Diptera, Drosophilidae) na área norte da “Cadeia do Espinhaço”, estados de Minas Gerais e Bahia, Brasil: aspectos biogeográficos e ecológicos. – Iheringia Ser. Zool. 78: 85 – 94.
- Tidon-Sklorz, R., Vilela, C. R., Sene, F. M. et al. 1994. O gênero Drosophila (Diptera, Drosophilidae) na Serra do Cipó, estado de Minas Gerais, Brasil. – Revta. Bras. Ent. 38: 627 – 637.
- Vilela, C. R., Sene, F. M. e Pereira, M. A. Q. R. 1980. Sobre a fauna de Drosophila do Chaco e das encostas orientais dos Andes na Argentina. – Revta. Bras. Biol. 40: 837 – 841. Vilela, C. R., Pereira, M. A. Q. R. e Sene, F. M. 1983. Dados preliminares sobre a distribuição geográfica de espécies de Drosophila dentro dos domínios morfoclimáticos do Brasil. II. O grupo repleta. – Ciên. Cult. São Paulo 35: 66 – 70.
- Wasserman, M. 1962. Estudos citológicos do grupo repleta do gênero Drosophila. V. O subgrupo mulleri. – Univ. Texas Publ. 6205: 85 – 118.
- Wu, W. L., Wang, J. P., Tseng, M. C. et al. 1999. Clonagem e variabilidade genética de um DNA repetitivo HindIII em Acrossocheilus paradoxus (Cyprinidae). – Genome 42: 780 – 788.