Baixa Variabilidade de DNA Satélite em Populações Naturais de Drosophila antonietae Envolvidas em Diferentes Eventos Evolutivos
Tradução automática
O artigo original está escrito em EN language (link para lê-lo) .
Drosophila antonietae é uma espécie cactofílica que é encontrada na floresta mesofílica da bacia do rio Paraná–Paraguai e nas dunas da costa atlântica sul do Brasil. Embora a estrutura genética das populações da bacia do rio Paraná–Paraguai já tenha sido estabelecida, a relação entre essas populações e aquelas na costa atlântica é controversa. Neste estudo, comparamos 33 unidades repetitivas do DNA satélite pBuM-2 isoladas de indivíduos de 8 populações de D. antonietae nessas regiões geográficas, incluindo algumas populações encontradas dentro de uma zona de contato com a estreitamente relacionada D. serido. As sequências de pBuM-2 mostraram baixa variabilidade interpoblacional. Este resultado foi interpretado como uma consequência tanto do fluxo gênico entre as populações quanto do crossing over desigual promovendo a homogeneização dos arranjos em tandem. Os resultados apresentados aqui, juntamente com os de estudos anteriores, destacam o uso de pBuM-2 para resolver conflitos taxonômicos dentro do grupo de espécies D. buzzatii.
Introdução
Drosophila antonietae é um membro do grupo de espécies D. buzzatii, um grupo monofilético composto por 7 espécies irmãs naturalmente endêmicas da região neotropical, que ocorrem em florestas abertas e secas encontradas no leste da América do Sul (Manfrin e Sene 2006). Essas espécies utilizam tecido necrosado de cactos como locais de reprodução (Pereira et al. 1983); por causa disso, sua distribuição geográfica é restrita à faixa de distribuição dos cactos hospedeiros. Assim, D. antonietae é um modelo atraente para estudar processos evolutivos (Manfrin e Sene 2006).
A distribuição geográfica de D. antonietae pode ser caracterizada de 2 maneiras. Nos vales enclausurados pela bacia do rio Paraná–Paraguai, populações são encontradas em florestas mesofílicas associadas ao cacto Cereus hildemannianus (Tidon-Sklorz e Sene 2001; Mateus e Sene 2003). Em sua distribuição sudeste, D. antonietae ocorre nas regiões de dunas da costa atlântica, utilizando principalmente C. hildemannianus, mas também cactos do gênero Opuntia (Ruiz et al. 2000; Manfrin e Sene 2006) (Figura 1).
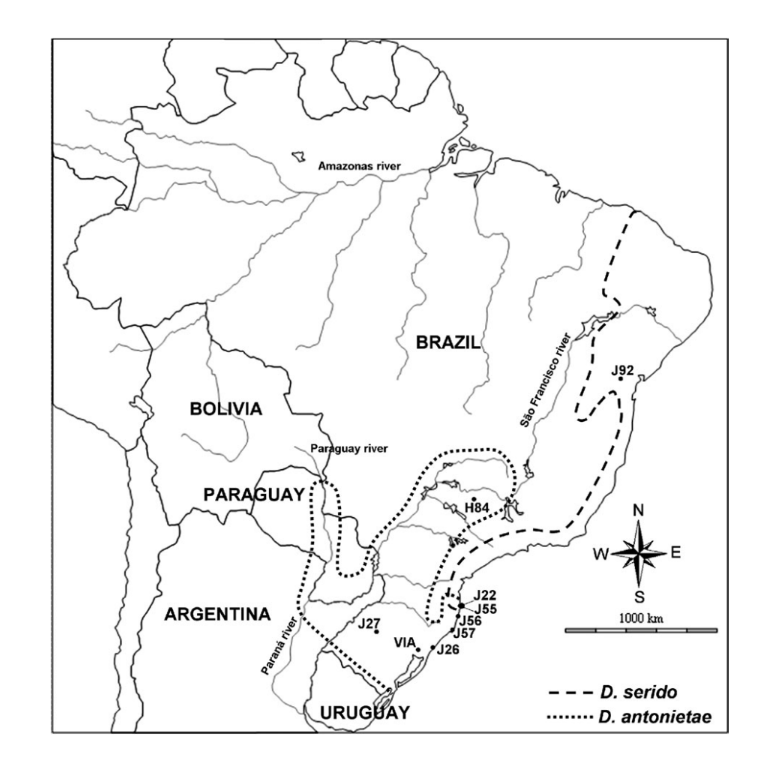
Estudos anteriores sugeriram que as populações de D. antonietae da bacia do rio Paraná–Paraguai estão estruturadas de acordo com o modelo de isolamento por distância devido à correlação positiva das distâncias morfológicas e genéticas com as distâncias geográficas e ecológicas (esta última definida como a distância entre 2 populações seguindo o curso do rio) (de Brito et al. 2002; Mateus e Sene 2007). Com base no número de migrantes por geração, Mateus e Sene (2007) sugeriram um efeito moderado tanto do fluxo gênico quanto da deriva genética nessas populações de D. antonietae, com o fluxo gênico sobrepondo a deriva genética promovendo um gradiente genético.
Uma análise filogeográfica baseada em DNA mitocondrial sugeriu que a distribuição sudeste de D. antonietae, na costa atlântica do Brasil, foi colonizada por eventos de expansão de populações de D. antonietae da depressão central do estado do Rio Grande do Sul (de Brito et al. 2002; Manfrin e Sene 2006). No entanto, essa análise não conseguiu determinar se essa expansão foi seguida pela manutenção do fluxo gênico, com aquisição de estrutura populacional de acordo com o modelo de isolamento por distância, ou seguida por fragmentação, implicando uma ausência de fluxo gênico entre as populações da costa atlântica e aquelas da bacia do rio Paraná–Paraguai (de Brito et al. 2002). Os dados de inversão cromossômica podem sugerir algum grau de fragmentação entre as populações desses 2 ambientes diferentes, uma vez que aquelas populações dos vales da bacia do rio Paraná–Paraguai são polimórficas para as inversões 2y8, 2z8 e 5e, enquanto as populações da costa atlântica são polimórficas para 2z8 (Ruiz et al. 2000). As populações da costa atlântica de D. antonietae têm outra característica importante: elas estão em contato com a espécie estreitamente relacionada D. serido (D. buzzatii cluster) (Ruiz et al. 2000; Manfrin e Sene 2006). Esta área de sobreposição é o resultado de D. serido se expandindo do norte para a costa atlântica sul e compartilhando um local geográfico com
D. antonietae populações (de Brito et al. 2002; Manfrin e Sene 2006) (Figura 1). Características morfológicas e análises de DNA mitocondrial sugerem que a hibridização entre
D. antonietae e D. serido ocorreu dentro da zona de contato (C.K.B. Santos e colegas, dados não publicados).
DNAs satélites são sequências de DNA altamente repetitivas organizadas em tandem que representam um componente substancial da heterocromatina de organismos eucarióticos (Charlesworth et al. 1tt4; Palomeque e Lorite 2008). Os satélites estão tipicamente localizados perto de regiões centroméricas e, com menos frequência, em telômeros (Ugarkovic e Plohl 2002; Kuhn et al. 2008). Essas sequências evoluem de maneira concertada, enquanto os principais mecanismos moleculares envolvidos em sua evolução são replicação por deslizamento, crossing over desigual, conversão gênica e replicação em círculo rolante (Dover 1982; Charlesworth et al. 1994).
Algumas das famílias de DNA satélite evoluem rapidamente, permitindo seu uso como marcadores moleculares em estudos comparativos interespecíficos (Watabe et al. 1997; Picariello et al. 2002; Kuhn e Sene 2005). No entanto, o uso dessas sequências como marcadores moleculares em estudos populacionais tem sido relativamente pouco explorado, apesar do fato de que a evolução concertada foi relatada em populações de Cyprinodon variegates (Cyprinodontidae) (Elder e Turner 1994) e Acrossocheilus paradoxus (Cyprinidae) (Wu et al. 1999).
Três famílias de DNA satélite compartilhadas entre as espécies do grupo D. buzzatii foram formalmente descritas: pBuM (Kuhn et al. 1999, 2008; Kuhn e Sene 2005), DBC-150 (Kuhn et al. 2007) e SSS139 (Franco et al. 2008). A família pBuM é composta por 2 subfamílias, pBuM-1 e pBuM-2 (Kuhn e Sene 2005; Kuhn et al. 2008). A subfamília pBuM-2 é composta por monômeros ricos em AT de 370 pb, que apresentam diferenças qualitativas (substituição de nucleotídeos fixos) e quantitativas (número de cópias) entre as espécies do grupo D. buzzatii (Kuhn e Sene 2005; Kuhn et al. 2007, 2008).
Neste trabalho, demonstramos a variabilidade intraespecífica do DNA satélite pBuM-2 em D. antonietae, utilizando populações de 2 ambientes diferentes (a bacia do Paraná–Paraguai e a costa atlântica) que apresentam histórias evolutivas distintas, associadas à manutenção do fluxo gênico no primeiro e eventos de hibridação com a espécie estreitamente relacionada D. serido no último. Escolhemos usar o DNA satélite pBuM como um marcador molecular porque ele apresenta 14 substituições nucleotídicas diagnósticas entre D. antonietae e D. serido (Kuhn e Sene 2005). Assim, o pBuM permite a identificação taxonômica de espécimes isolados da zona de contato. Além disso, como a dinâmica evolutiva das sequências de DNA satélite no nível intraespecífico não é completamente compreendida, dados empíricos adicionais podem contribuir para uma melhor compreensão do processo subjacente à evolução do DNA satélite (Ugarkovic e Plohl 2002; Palomeque e Lorite 2008).
Material e Métodos
Amostras
Estudamos 8 populações brasileiras de D. antonietae (Figura 1). Três dessas populações estão localizadas nos vales da bacia do rio Paraná–Paraguai: J2/ (Santiago, estado do Rio Grande do Sul), Via (Viamão, estado do Rio Grande do Sul) e H84 (Serrana, estado de São Paulo, número de acesso AY656609– AY656615; Kuhn e Sene 2005); e 5 estão localizadas nas dunas da costa atlântica, dentro da zona de contato com
D. serido: J56 (Garopaba, estado de Santa Catarina), J55 (praia de Armaxcão, Florianópolis, estado de Santa Catarina), J22 (praia Joaquina, Florianópolis, estado de Santa Catarina), J5/ (Laguna, estado de Santa Catarina) e J26 (Osório, estado do Rio Grande do Sul) (Figura 1). As sequências de D. antonietae foram analisadas juntamente com sequências de G pBuM-2 de D. serido (população Jf2—Milagres, estado da Bahia, número de acesso AY656616–AY656621; Kuhn e Sene 2005), a fim de identificar alguns eventos de introgressão em populações da costa atlântica.
Isolamento do DNA Satélite pBuM-2 e Análise de Sequência
O DNA genômico de um indivíduo macho (moscas capturadas na natureza) por população foi extraído usando o Kit de Purificação de DNA Genômico Wizard (Promega). O DNA genômico foi submetido à reação em cadeia da polimerase para amplificar as cópias da família pBuM-2 [primers A2F (CGGAGTA-TTTTTCATTCGAC) e A2R (GGTATGCCATAAAG-AAGTCG)] de acordo com Kuhn et al. (2008). As bandas resultantes de aproximadamente 400 bp foram eluídas do gel por incubação noturna em uma solução de eluição (500 mM NaAc; 1 mM ácido etilenodiaminotetraacético). Os fragmentos recuperados foram clonados usando o kit de clonagem de extremidade cega pMOSBlue (RPN 5110; Amersham Pharmacia Biotech). O DNA plasmidial foi preparado seguindo a metodologia descrita em Sambrook et al. (1989), e a reação de template de DNA para sequenciamento foi preparada de acordo com o manual do Kit de Reação Pronta de Sequenciamento por Ciclo BigDye Terminator (PerkinElmer). O sequenciamento automático de DNA foi realizado em um sequenciador ABI Prism 377 (PerkinElmer).
Os alinhamentos de sequência foram realizados no CLUSTALW 1.8 (Thompson et al. 1994) e editados no BioEdit (Hall 1999). Usamos o algoritmo e o modelo estatístico proposto por Betrán et al. (1997) para detectar trechos de conversão gênica. O parâmetro Rm, que estima o número mínimo de eventos de recombinação em uma amostra de DNA (Hudson e Kaplan 1985), foi utilizado para detectar eventos de recombinação. Embora esse parâmetro não meça especificamente a troca desigual, é razoável supor que o Rm teria valores superiores a zero se a troca desigual tivesse ocorrido. Nesse sentido, usamos Rm como evidência indireta de troca desigual. As distâncias genéticas foram calculadas de acordo com a p-distância, que é a proporção de diferenças nucleotídicas entre 2 sequências (ou seja, p 5 Np/N, onde Np são as diferenças de nucleotídeos e N é o número total de nucleotídeos). Modelos de distância mais complexos também foram aplicados aos dados, com resultados semelhantes aos apresentados pela p-distância (dados não mostrados). A matriz de distância foi utilizada para criar um fenograma usando o método de vizinho mais próximo (Saitou e Nei 1987). Os cálculos de distância genética e fenogramas foram feitos usando o programa MEGA 3.0 (Kumar et al. 2004).
Também investigamos padrões de variação intraespecífica em D. antonietae por meio de análises de variância molecular (AMOVAs; Excoffier et al. 1992). Para a AMOVA, as populações de D. antonietae foram agrupadas em 2 grupos: costa atlântica e bacia do rio Paraná–Paraguai. A AMOVA foi realizada usando Arlequin 3.0 (Excoffier et al. 2005).
Resultados e Discussão
Vinte e seis novos monômeros de DNA satélite pBuM-2 foram obtidos: 4 da localidade J56 (número de acesso FJ935973– FJ93597G); 4 da localidade Via (número de acesso FJt35t77–FJt35t80); 4 da localidade J26 (número de acesso FJ935981–FJ935984); 3 da localidade J5/ (número de acesso FJ935985–FJ935987); 5 da localidade J2/ (número de acesso FJ935988–FJ935992); 2 da localidade J55 (número de acesso FJ935993–FJ935994); e 4 da localidade J22 (número de acesso FJ935995–FJ935998). A alta similaridade nucleotídica entre as sequências obtidas neste trabalho e aquelas previamente descritas (Kuhn e Sene 2005) confirma que pertencem à família de DNA satélite pBuM-2.
Todas as sequências obtidas apresentam a estrutura primária típica de D. antonietae e foram agrupadas no mesmo ramo no fenograma, juntamente com as sequências pBuM-2 previamente descritas de D. antonietae (Figura 2), mesmo aquelas das populações dentro da zona de contato com D. serido, onde foi sugerida a hibridização entre D. antonietae e D. serido com introgressão de DNA mitocondrial (C.K.B Santos, F.M. Sene e M.H. Manfrin, dados não publicados). Essa situação pode estar relacionada às observações de que os genes mitocondriais tendem a introgressar mais facilmente do que as sequências nucleares (Dorado et al. 1992; Arnold 1993). As explicações mais citadas para essa observação estão relacionadas a processos de desequilíbrios cito-nucleares (Arnold 1993) e à esterilidade de machos híbridos (Aubert e Solignac 1990).
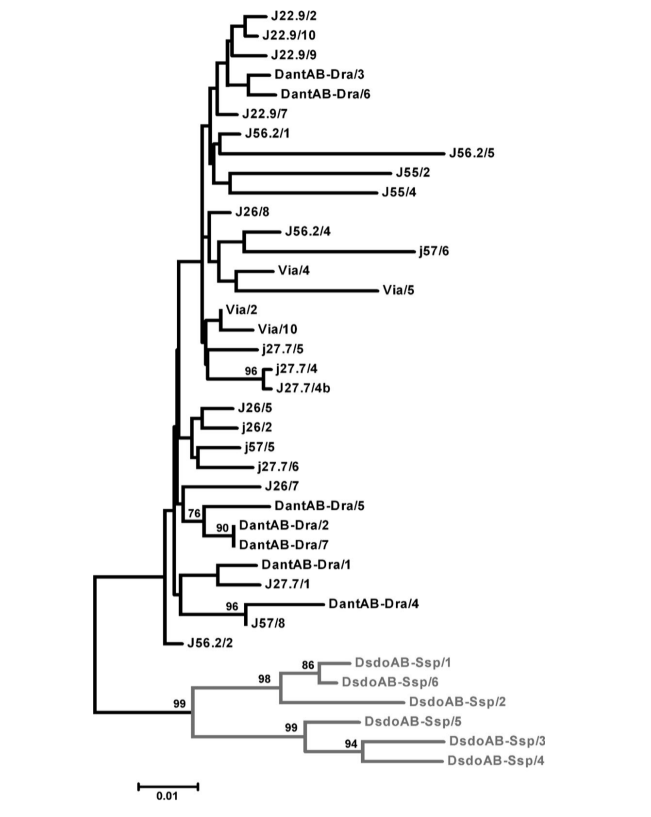
Há um alto nível de similaridade de sequência entre as sequências de pBuM-2 de D. antonietae (95,7% em média), e nenhuma mutação foi encontrada como diagnóstica para uma população específica. A variabilidade interpoblacional foi muito semelhante à variabilidade intrapoblacional (Tabela 1), e sequências da mesma população não se agruparam juntas em um ramo específico no dendrograma (Figura 2). A AMOVA realizada com as populações subdivididas por grupos geográficos (bacia do rio Paraná–Paraguai e costa atlântica) mostrou que 99,30% da diversidade genética total poderia ser atribuída à variabilidade intragrupo, enquanto apenas 0,70% foi atribuída à variabilidade intergrupo (Tabela 2). O Фst calculado foi baixo e não teve suporte estatístico (Tabela 2).
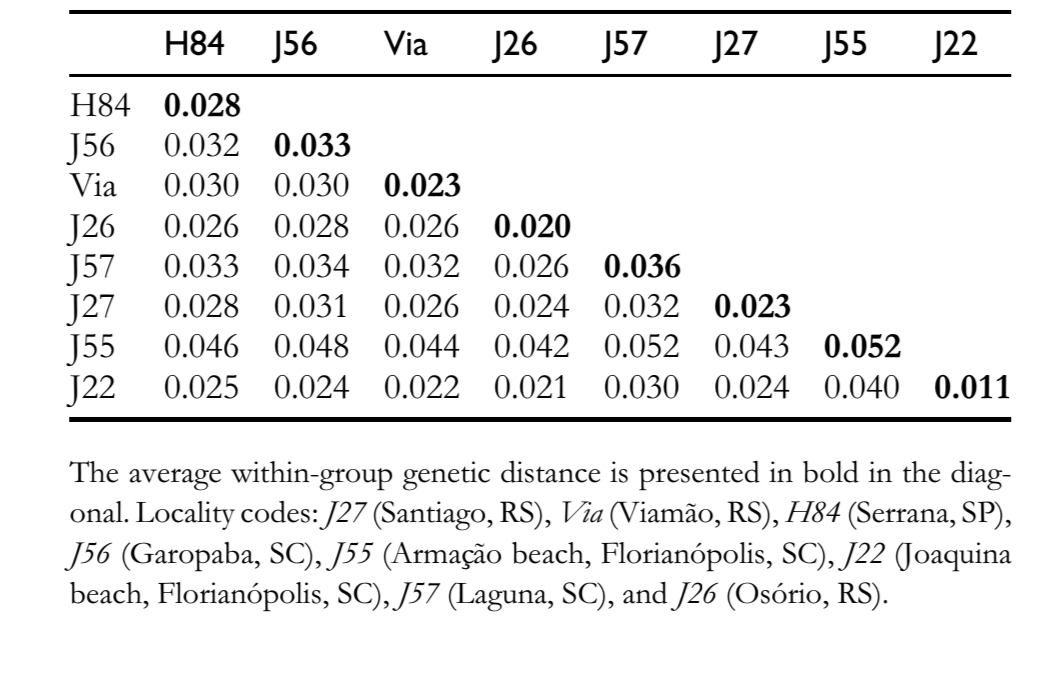

As populações de D. antonietae da bacia do rio Paraná–Paraguai estão estruturadas de acordo com um modelo de isolamento por distância (Monteiro e Sene 1995; Mateus e Sene 2007). As inversões cromossômicas observadas sugerem fragmentação entre as populações da costa atlântica e aquelas da bacia do rio Paraná–Paraguai (Ruiz et al. 2000), enquanto os dados de DNA mitocondrial não puderam concluir se a fragmentação ou o isolamento por distância era a estrutura dominante definindo a relação desses 2 grupos populacionais (de Brito et al. 2002). A alta similaridade nucleotídica de pBuM-2 em D. antonietae (Figura 2 e Tabelas 1 e 2) é congruente com a manutenção do fluxo gênico entre as populações dessa espécie, mesmo entre aquelas alocadas na bacia do rio Paraná–Paraguai e na costa atlântica. Nossos resultados sugerem que o isolamento por distância deve explicar a variabilidade do DNA mitocondrial em D. antonietae; este modelo prevê fluxo gênico entre as populações, conforme inferido com base nos dados de DNA satélite presentes aqui. Nesse contexto, os menores polimorfismos de inversão cromossômica nas populações da costa atlântica de D. antonietae, detectados por Ruiz et al. (2000), podem ser explicados como uma consequência de eventos históricos e demográficos, como a expansão populacional a partir das populações dos vales da bacia do rio Paraná–Paraguai, conforme sugerido pelo DNA mitocondrial (de Brito et al. 2002). Alternativamente, essa diferenciação populacional pode estar associada à adaptação local a ambientes distintos na presença de fluxo gênico, uma vez que as inversões cromossômicas podem estar correlacionadas com características adaptativas em Drosophila cactofílicas (Hasson et al. 1992; Fernández Iriarte et al. 2003). Uma explicação alternativa para a alta conservação nucleotídica de pBuM-2 em D. antonietae é baseada na dinâmica evolutiva das sequências de DNA satélite. Mecanismos moleculares relacionados à homogeneização de sequências dispostas em tandem, como crossing over desigual e conversão gênica através de um processo chamado de "molecular drive" (Dover 1982; Strachan et al. 1985), poderiam estar atuando no DNA satélite pBuM nesta espécie. Nossas análises não identificaram traços de conversão gênica nas sequências de pBuM-2 isoladas de D. antonietae, sugerindo que esse mecanismo molecular não influencia a diversidade de sequências de pBuM-2 nesta espécie. No entanto, um mínimo de 4 eventos de recombinação foi detectado na amostra (Rm = 4) entre os intervalos de 130–136, 136–155, 174–376 e 376–378. Esses eventos de recombinação poderiam ser uma evidência indireta de crossing over desigual contribuindo para a manutenção da estrutura primária das sequências de pBuM-2 nas diferentes populações de D. antonietae. Consistente com essa hipótese, o genoma de D. antonietae contém um grande número de monômeros de pBuM-2 (Kuhn et al. 2007; Kuhn et al. 2008), fato que aumenta a probabilidade de crossing over desigual (Smith1976), e consequentemente homogeneização e evolução concertada.
Essas 2 hipóteses alternativas que explicam a alta similaridade nucleotídica encontrada nos monômeros de pBuM-2 de D. antonietae não são mutuamente exclusivas. Nesse contexto, propomos que um equilíbrio entre o fluxo gênico entre populações e eventos de homogeneização dos arranjos em tandem do DNA satélite, promovidos pelo crossing over desigual, contribui para a conservação da estrutura primária do pBuM-2 nas espécies de D. antonietae.
A ausência de divergência molecular nas sequências de DNA satélite entre populações da mesma espécie, como encontrado no presente artigo, também foi observada em outras espécies de insetos (Bachmann et al. 1994, 1998; Lorite et al. 2002; Feliciello et al. 2005). Por exemplo, Bachmann et al. (1994) encontraram altos níveis de similaridade genética entre monômeros do pDoP102 satélite em populações de Dolichopoda schiavazzii (Orthoptera), cujos dados alozimáticos indicam que estiveram isolados uns dos outros por pelo menos 235 000 anos. Segundo esses autores, a homogeneidade em pDoP102 mesmo em populações isoladas poderia ser devido à biologia do DNA satélite, pois o crossing over desigual e a conversão gênica podem induzir a manutenção da estrutura primária das sequências de DNA satélite (Bachmann et al. 1994). Em outro estudo, a presença de trechos de conversão gênica nos monômeros de uma família de DNA satélite foi usada como evidência de eventos de homogeneização nesse DNA satélite, explicando a alta conservação intraspecífica do DNA satélite encontrada no besouro fitófago Xanthogaleruca luteola (Lorite et al. 2002).
No grupo de espécies D. buzzatii, uma alta conservação intraespecífica da sequência pBuM-2 também foi identificada em D. seriema (Kuhn e Sene 2004) e D. gouveai (Franco, Kuhn, et al. 2006). Além disso, as populações de D. buzzatii são homogêneas em relação ao DNA satélite pBuM-1, outro membro da família pBuM (Kuhn et al. 2003). Em D. seriema e D. buzzatii, a falta de diferenciação populacional nas sequências de DNA satélite foi sugerida como resultado de eventos de fluxo gênico passados e atuais entre populações, que são compatíveis com os resultados obtidos de outros marcadores genéticos analisados nas mesmas populações (Kuhn et al. 2003; Kuhn e Sene 2004). Para
D. gouveai, que é estruturado de acordo com um modelo de ‘‘ilha’’ (de Brito et al. 2002; Moraes e Sene 2007), a principal hipótese para explicar a conservação da estrutura primária no DNA satélite pBuM-2 foi que mecanismos de homogeneização independentes que favorecem unidades repetitivas compartilhadas entre as populações ocorreram após o evento de isolamento das populações de D. gouveai (Franco, Kuhn, et al. 2006). O grupo D. buzzatii é composto por espécies irmãs com taxonomia baseada nas características morfométricas do aedeago, que é o órgão intromitente dos genitais masculinos dos insetos (Vilela 1983; Tidon-Sklorz e Sene 2001; Franco, Prado, et al. 2006). Assim, a identificação de indivíduos fêmeas do grupo D. buzzatii, provenientes do ambiente natural, é feita pela identificação de sua prole masculina, tornando necessário o estabelecimento de linhas isofêmeas, o que pode ser uma tarefa difícil para espécies de Drosophila cactofílicas. Além disso, a discriminação correta das espécies baseada apenas em caracteres quantitativos, como a morfologia do aedeago, às vezes é difícil devido à presença de indivíduos morfologicamente ambíguos, especialmente quando os indivíduos são coletados em zonas de contato entre espécies do grupo D. buzzatii. Nesse sentido, a forte conservação intraespecífica das sequências de DNA satélite pBuM obtidas em vários estudos (Kuhn et al. 2003; Kuhn e Sene 2004; Franco, Kuhn, et al. 2006; artigo presente), mesmo em espécies geneticamente estruturadas, destaca a utilidade do pBuM-2 na resolução de conflitos taxonômicos dentro das espécies do grupo D. buzzatii, um modelo reconhecido para estudos de biologia evolutiva (Manfrin e Sene 2006).
Autores: Fernando Faria Franco, Fabio Melo Sene, Maura Helena Manfrin
Referências:
- Arnold J. 1993. Desequilíbrios cito-nucleares em zonas híbridas. Annu Rev Ecol Syst. 24:521–554.
- Aubert J, Solignac M. 1990. Evidência experimental de introgressão de DNA mitocondrial entre espécies de Drosophila. Evolution. 44:1272–1282.
- Bachmann L, Tomiuk J, Adis J, Vohland K. 1998. Diferenciação genética do milípede Pycnotropis epiclysmus que habita florestas amazônicas sazonalmente inundadas e não inundadas. J Zool Syst Evol Res. 36:65–70.
- Bachmann L, Venanzetti F, Sbordoni V. 1994. Caracterização de uma família de DNA satélite específica de espécie de Dolichopoda schiavazzi (Orthoptera, Rhaphidophoridae) grilos de caverna. J Mol Evol. 39:274–281.
- Betrán E, Rozas J, Navarro A, Barbadilla A. 1997. A estimativa do número e da distribuição de comprimento dos trechos de conversão gênica a partir de dados de sequência de DNA populacional. Genetics. 146:89–99.
- Charlesworth B, Sniegowski P, Stephan W. 1tt4. A dinâmica evolutiva do DNA repetitivo em eucariotos. Nature. 371:215–220.
- de Brito AR, Manfrin MH, Sene FM. 2002. Análise cladística aninhada de populações brasileiras de Drosophila serido. Mol Phylogenet Evol. 22: 131–143.
- Dorado O, Rieseberg LH, Arias D. 1992. Introgressão de DNA de cloroplasto em girassóis do sul da Califórnia. Evolution. 46:566–572.
- Dover G. 1982. Impulso molecular: um modo coeso de evolução das espécies. Nature. 299:111–117.
- Elder JF, Turner BJ. 1tt4. Evolução concertada a nível populacional: sequências de DNA satélite HindIII de peixes-pupa. Proc Natl Acad Sci U S A. 91: 994–998.
- Excoffier L, Laval G, Schneider S. 2005. Arlequin (versão 3.0): um pacote de software integrado para análise de dados de genética populacional. Evol Bioinform Online. 1:47–50.
- Excoffier L, Smouse PE, Quattro JM. 1992. Análises de variância molecular inferidas a partir de distâncias métricas entre haplótipos de DNA: aplicação a dados de restrição de DNA mitocondrial humano. Genetics. 131:479–491.
- Feliciello I, Picariello O, Chinali G. 2005. A primeira caracterização da variabilidade geral de unidades repetitivas em uma espécie revela características inesperadas do DNA satélite. Gene. 349:153–164.
- Fernández Iriarte PJ, Norry FM, Hasson ER. 2003. Inversões cromossômicas afetam o tamanho e a forma do corpo em diferentes recursos de reprodução em Drosophila buzzatii. Heredity. 91:51–59.
- Franco FF, Kuhn GCS, Sene FM, Manfrin MH. 2006. Conservação das sequências de DNA satélite pBuM–2 entre populações geograficamente isoladas de Drosophila gouveai do Brasil. Genetica. 128:287–295.
- Franco FF, Prado PRR, Sene FM, Costa LF, Manfrin MH. 200G. Morfologia do aedeago como um marcador discriminante em duas espécies cactofílicas intimamente relacionadas de Drosophila (Diptera; Drosophilidae) na América do Sul. An Acad Bras Cienc. 78:203–212.
- Franco FF, Sene FM, Manfrin MH. 2008. Caracterização molecular da família de DNA satélite SSS13t em espécies irmãs do grupo Drosophila buzzatii. Genet Mol Biol. 31:155–159.
- Hall TA. 1999. BioEdit: um editor de alinhamento de sequências biológicas amigável ao usuário e programa de análise para Windows t5/t8/NT. Nucleic Acids Symp Ser. 41:95–98.
- Hasson E, Fanara JJ, Rodríguez C, Vilardi JC, Reig OA, Fontdevila A. 1992. A história evolutiva de Drosophila buzzatii. XXIV. Inversões do segundo cromossomo têm efeito médio diferente sobre o comprimento do tórax. Heredity. 68:557–563.
- Hudson RR, Kaplan NL. 1985. Propriedades estatísticas do número de eventos de recombinação na história de uma amostra de sequências de DNA. Genetics. 111:147–164.
- Kuhn GCS, Bollgönn S, Sperlich D, Bachmann L. 1999. Caracterização de um DNA satélite específico de espécie de Drosophila buzzatii. J Zool Syst Evol Res. 37:109–112.
- Kuhn GCS, Franco FF, Manfrin MH, Moreira-Filho O, Sene FM. 2007. Baixas taxas de homogeneização da família de DNA satélite DBC-150 restrita a um único par de microcromossomos em espécies do grupo Drosophila buzzatii. Chromosome Res. 15:457–469.
- Kuhn GCS, Franco FF, Silva WA Jr, Martinez-Rossi NM, Sene FM. 2003. Sobre a variabilidade do DNA satélite pBuM18t entre populações sul-americanas de Drosophila buzzatii. Hereditas. 139:161–166.
- Kuhn GCS, Sene FM. 2004. Caracterização e variabilidade interpoblacional de um DNA satélite complexo HpaI de Drosophila seriema (grupo repleta). Genetica. 121:241–249.
- Kuhn GCS, Sene FM. 2005. Rotatividade evolutiva de duas subfamílias de DNA satélite pBuM no grupo de espécies Drosophila buzzatii (grupo repleta): de alpha para alpha/beta arrays. Gene. 349:77–85.
- Kuhn GCS, Sene FM, Moreira-Filho O, Schwarzacher T, Heslop-Harrison JS. 2008. Análise de sequência, distribuição cromossômica e organização de longo alcance mostram que a rápida rotatividade de novos e antigos repetições de DNA satélite pBuM leva a diferentes padrões de variação em sete espécies do grupo Drosophila buzzatii. Chromosome Res. 16:307–324.
- Kumar S, Tamura K, Nei M. 2004. MEGA3: software integrado para análise de genética evolutiva molecular e alinhamento de sequências. Brief Bioinform. 5:150–163.
- Lorite P, Carrillo JA, Garneria I, Petitpierre E, Palomeque T. 2002. DNA satélite no besouro da folha do olmo, Xanthogaleruca luteola (Coleoptera, Chrysomelidae): caracterização, análise interpoblacional e localização cromossômica. Cytogenet Genome Res. 98:302–307.
- Manfrin MH, Sene FM. 200G. Cactofílicos Drosophila na América do Sul: um modelo para estudos evolutivos. Genetica. 126:57–75.
- Mateus RP, Sene FM. 2003. Variação alozima temporal e espacial na cactofílica Drosophila antonietae (Diptera; Drosophilidae) da América do Sul. Biochem Genet. 41:219–233.
- Mateus RP, Sene FM. 2007. Estudo genético populacional da variação alozima em populações naturais de Drosophila antonietae (Insecta, Diptera). J Zool Syst Evol Res. 45:136–143.
- Monteiro SG, Sene FM. 1tt5. Estudo morfométrico de populações de Drosophila serido das regiões Central e Sul do Brasil. Rev Bras Genet (Suppl). 18:283.
- Moraes EM, Sene FM. 2007. Variação de microssatélites e morfométrica em Drosophila gouveai: a importância relativa de fatores históricos e atuais na formação da estrutura genética populacional. J Zoolog Syst Evol Res. 45:336–344.
- Palomeque T, Lorite P. 2008. DNA satélite em insetos: uma revisão. Heredity. 100:564–573.
- Pereira MAQR, Vilela CR, Sene FM. 1983. Notas sobre locais de reprodução e alimentação de algumas espécies do grupo repleta do gênero Drosophila (Diptera, Drosophilidae). Cienc Cult. 35:1313–1319.
- Picariello O, Feliciello I, Bellinero R, Chinali G. 2002. DNA satélite S1 como um marcador taxonômico em rãs marrons: evidência molecular de que Rana graeca graeca e Rana graeca italica são espécies diferentes. Genome. 45: 63–70.
- Ruiz A, Cassian AM, Kuhn GCS, Alves MAR, Sene FM. 2000. O quebra-cabeça da especiação de Drosophila serido: juntando novas peças. Genetica. 108:217–227.
- Saitou N, Nei M. 1987. O método de vizinho-união: um novo método para reconstruir árvores filogenéticas. Mol Biol Evol. 4:406–425.
- Sambrook J, Fritsh EF, Maniatis T. 1989. Clonagem Molecular: um manual de laboratório. 2ª ed. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press.
- Smith GP. 1976. Evolução de sequências de DNA repetidas por crossover desigual. Science. 191:528–535.
- Strachan T, Webb D, Dover GA. 1985. Estágios de transição do impulso molecular em famílias de DNA de cópias múltiplas em Drosophila. EMBO J. 4:1701–1708.
- Thompson JD, Higgins DG, Gibson TJ. 1994. CLUSTAL W: melhorando a sensibilidade do alinhamento progressivo de múltiplas sequências através de ponderação de sequências, penalidades de lacunas específicas de posições e escolha de matriz de pesos. Nucleic Acids Res. 22:4673–4680.
- Tidon-Sklorz R, Sene FM. 2001. Duas novas espécies do conjunto de irmãos de Drosophila serido (Diptera, Drosophilidae). Iheringia. 90:141–146.
- Ugarkovic D, Plohl M. 2002. Variação nos perfis de DNA satélite—causas e efeitos. EMBO J. 21:5955–5959.
- Vilela CR. 1983. Uma revisão do grupo de espécies Drosophila repleta (Diptera; Drosophilidae). Rev Bras Entomol. 27:1–114.
- Watabe H, Bachmann L, Haring E, Sperlich D. 1997. Estudos taxonômicos e moleculares sobre Drosophila sinobscura e D. hubeiensis, duas espécies irmãs do grupo D. obscura. J Zool Syst Evol Res. 35:81–94.
- Wu WL, Wang JP, Tseng MC, Chiang TY. 1999. Clonagem e variabilidade genética de um DNA repetitivo HindIII em Acrossocheilus paradoxos (Cyprinidae). Genome. 42:780–788.
